Genetische genealogie, een nieuw onderzoeksinstrument in onopgeloste strafzaken

Noorse forensisch wetenschappers rapporteren dat ze genetische genealogie hebben gebruikt om drie onopgeloste strafrechtelijke onderzoeken vooruit te helpen. In twee gevallen identificeerde de techniek met succes de persoon wiens DNA op de plaats delict werd gevonden. Volgens de auteurs van de studie, gepubliceerd in maart 2025 in het tijdschrift Forensic Science International: Genetics , tonen deze resultaten aan dat de aanpak voldoende volwassen is om een onbekende persoon te identificeren aan de hand van DNA dat op een plaats delict is aangetroffen.
Om een dergelijke methode in een politieonderzoek te kunnen gebruiken, is het noodzakelijk om over een biologisch monster te beschikken dat een bruikbare hoeveelheid DNA bevat. Het is ook noodzakelijk dat het biologische spoor ondubbelzinnig aan het misdrijf kan worden gekoppeld. Deze beoordeling moet worden uitgevoerd door de politie, met ondersteuning van forensisch deskundigen.
Hoewel genetische genealogie voor het eerst begin 2018 werd gebruikt, werd het een paar maanden later breed bekend met de identificatie van de Golden State Killer. Deze voormalige politieagent, Joseph James DeAngelo, werd niet geïdentificeerd aan de hand van zijn eigen DNA, maar aan de hand van dat van familieleden. Een achterneef uploadde zijn genetisch profiel naar de Californische website GEDmatch, waardoor het onderzoek naar hem kon worden herleid. DeAngelo, de dader van tientallen moorden, verkrachtingen en inbraken in Californië in de jaren 70 en 80, was al 42 jaar vermist.
Sinds de arrestatie van Joseph DeAngelo in april 2018 heeft genetische genealogie, toegepast in strafrechtelijk onderzoek, aanzienlijke belangstelling gewekt en zich bewezen als een bijzonder effectief instrument voor het oplossen van zaken die soms al tientallen jaren onopgelost zijn. Dit keerpunt wordt deels verklaard door het opvallende contrast tussen de middelen die vóór de invoering van deze methode werden ingezet en de effectiviteit ervan toen deze eenmaal was geïmplementeerd: het duurde vier decennia, 650 rechercheurs, vijftien verschillende politiediensten en bijna 10 miljoen dollar voordat DeAngelo's DNA eindelijk in de GEDmatch-database werd geüpload. Na deze stap waren er slechts vijf genealogen, een paar honderd dollar en amper 63 dagen nodig om de verdachte te identificeren.
In Europa dateert het eerste gedocumenteerde gebruik van deze methode uit 2019, in Zweden, om een dubbele moord op te lossen die 15 jaar eerder in Linköping was gepleegd. In 2023 gebruikte de Australische politie de methode om twee cold cases op te lossen. Datzelfde jaar startte Nederland een pilotstudie met twee onopgeloste zaken. In 2024 gebruikte Nieuw-Zeeland de methode in twee moordzaken die teruggingen tot 1980 en 2008.
Tegenwoordig bestuderen andere landen, waaronder het Verenigd Koninkrijk, Zweden, Australië, China en Spanje, forensisch genetisch genealogisch onderzoek of hebben dit al geïmplementeerd.
In Frankrijk werd in 2022 Bruno L., een 62-jarige gepensioneerde woonachtig in Seine-et-Marne, door genetische genealogie geïdentificeerd als de dader van een reeks verkrachtingen gepleegd tussen 1998 en 2008 in de regio Parijs. Ondanks de herhaaldelijke aanwezigheid van zijn DNA op plaats delict, werd zijn profiel niet opgenomen in het Nationaal Geautomatiseerd Genetisch Vingerafdrukbestand (FNAEG). Frankrijk vroeg de FBI om hulp bij het uitvoeren van analyses via buitenlandse databanken. Er werden twee verre familieleden gevonden, waardoor de persoon naar hem kon worden herleid, die de feiten bekende voordat hij in de gevangenis zelfmoord pleegde.
Tot op heden zijn ongeveer 500 zaken opgelost met behulp van forensische genetische genealogie. Deze methode heeft geholpen bij het oplossen van lang onopgeloste geweldsmisdrijven, het identificeren van anonieme menselijke resten en zelfs het vrijpleiten van ten minste twee personen.
Momenteel staan drie genetische genealogische databases (GEDmatch PRO, FamilyTreeDNA, DNASolves) expliciet het uploaden van genetische gegevensbestanden van biologische monsters toe ten behoeve van genetische genealogie ten behoeve van strafrechtelijk onderzoek.
Zoek naar personen die verwant zijn aan de daderDe persoon aan wie dit DNA wordt toegeschreven, wordt het 'target' genoemd. In veel gevallen blijkt dit target de dader te zijn, maar breder gezien kan het iedereen zijn, levend of overleden, die van belang kan zijn voor het onderzoek.
De drie zaken waaraan Håvard Aanes en zijn collega's van de Universiteit van Oslo werkten, betroffen verkrachting en moord. In beide gevallen beschikte de politie over een bruikbaar DNA-profiel, maar dat had nog niet geleid tot de identificatie van verdachten.
In het eerste geval zijn rechercheurs op zoek naar de dader van twee verkrachtingen die vlak bij elkaar plaatsvonden. Een spermamonster dat op een van de locaties was verzameld, werd geanalyseerd: het bevatte DNA met een concentratie van 0,4 nanogram per microliter, oftewel 0,4 miljardste gram per miljoenste liter!
De tweede zaak betreft de verkrachting van een jonge vrouw, die plaatsvond tijdens een grote diploma-uitreiking van enkele duizenden middelbare scholieren. Het in deze zaak geanalyseerde spermamonster bevatte DNA in een hogere concentratie: 8,4 nanogram per microliter.
Het derde misdrijf dateert uit de zomer van 1999. Het lichaam van een gepensioneerde taalkundige werd twee weken na zijn dood gevonden in zijn appartement in het centrum van Oslo. De omstandigheden van het overlijden, gecombineerd met de tijd die was verstreken sinds de ontdekking, compliceerden het onderzoek, waardoor deze zaak een van de weinige onopgeloste moorden in Noorwegen was. Ter plaatse namen forensisch onderzoekers epitheelcellen af (van de huid of slijmvliezen). Met dit ene monster kon DNA worden geëxtraheerd met een concentratie van 0,2 nanogram per microliter.
DNA, genetische profilering en forensisch onderzoekOm te begrijpen hoe genetische genealogie wordt gebruikt voor forensische doeleinden bij politieonderzoeken om een verdachte te identificeren, is het eerst nodig om te begrijpen dat het basisidee is om DNA dat op een plaats delict wordt gevonden, niet te gebruiken om dit rechtstreeks naar de dader te herleiden, maar naar mensen die verwant zijn aan hem.
Voordat we kijken hoe DNA verwantschap kan aantonen, moeten we eerst bekijken hoe genetisch materiaal van generatie op generatie wordt doorgegeven.
Ons genetisch materiaal bestaat uit 23 paar chromosomen, oftewel 46 chromosomen in totaal. Deze chromosomen zijn lange DNA-moleculen. Hoewel alle mensen meer dan 99% van hun DNA delen, zijn het de resterende kleine verschillen, genetische variaties genaamd, die ons van elkaar onderscheiden en ons in staat stellen verwantschapsrelaties aan te gaan.
Op deze chromosomen variëren bepaalde specifieke posities van individu tot individu. De meest voorkomende en meest bruikbare voor genealogische analyse zijn SNP's (uitgesproken als "snips"), een afkorting voor Single Nucleotide Polymorphisms .
Dit zijn specifieke posities in het DNA waar één letter (A, T, C of G) van persoon tot persoon verschilt. Zo kan de ene persoon op een bepaalde locatie een A hebben en de ander een G. Deze variatiepunten, die overal in het genoom aanwezig zijn, dienen als kleine ijkpunten voor vergelijking tussen individuen.
Deze variaties (ook wel genetische polymorfismen genoemd) zijn enorm talrijk: er zijn er enkele miljoenen in het menselijk genoom. Ze zijn waardevol voor genetische genealogie, omdat ze ons in staat stellen het genoom van twee mensen te vergelijken en identieke segmenten te identificeren die door een gemeenschappelijke voorouder zijn overgedragen.
Om te begrijpen hoe deze DNA-segmenten op identieke wijze worden doorgegeven of juist opnieuw worden bewerkt over generaties heen, moeten we twee fundamentele begrippen introduceren: meiose en genetische recombinatie.
Meiose en genetische recombinatieTijdens de vorming van geslachtscellen (sperma en eicellen) vindt een speciaal proces plaats: meiose.
Meiose is een specifiek celdelingsmechanisme dat geen simpele kopieën van ouderlijk DNA produceert, maar unieke combinaties genereert. Waarom? Omdat tijdens dit proces homologe chromosomen van vaderlijke en moederlijke oorsprong paren vormen en delen van DNA met elkaar uitwisselen. Dit fenomeen, genetische recombinatie (of crossing-over ) genoemd, draagt bij aan genetische vermenging.
Deze recombinatie tussen homologe chromosomen is essentieel voor het goede verloop van de meiose. Het maakt het mogelijk om genen te herverdelen tussen de chromosomen die van de vader zijn geërfd en die van de moeder, waardoor originele associaties ontstaan. Dankzij deze vermenging bevat elke zaadcel of eicel een unieke samenstelling van genetisch materiaal. Met andere woorden, de twee chromosomen van hetzelfde paar (één van vaderskant, de andere van moederskant) kunnen DNA-fragmenten met elkaar uitwisselen. Dit proces omvat precieze breuken in de DNA-moleculen, gevolgd door reparaties die de uitgewisselde segmenten weer aan elkaar hechten. Deze recombinaties vinden plaats op verschillende plaatsen op elk chromosomenpaar.
Het resultaat? Elke voortplantingscel draagt een set recombinante chromosomen – een unieke mix van vaderlijke en moederlijke segmenten. Twee broers en zussen ontvangen dus elk een andere combinatie van het DNA van hun ouders: gemiddeld delen ze 50% van hun genetisch materiaal, maar niet exact dezelfde segmenten.
Dit mechanisme heeft directe gevolgen voor de analyse van genetische verwantschap. Wanneer we het DNA van twee mensen vergelijken om te bepalen of ze een gemeenschappelijke voorouder delen, zoeken we naar segmenten die identiek zijn door afstamming: delen van het DNA die zonder onderbreking zijn overgedragen van deze gemeenschappelijke voorouder. Maar hoe groter het aantal generaties tussen de twee individuen, hoe meer deze initiële segmenten door opeenvolgende recombinaties zijn gefragmenteerd.
Dit proces van recombinatie, essentieel voor genetische diversiteit, heeft een belangrijk gevolg voor de analyse van verwantschap: het fragmenteert geleidelijk de segmenten die van een voorouder zijn geërfd. Zo delen neven en nichten nog steeds lange DNA-segmenten. Daarentegen delen twee mensen met een gemeenschappelijke voorouder die vier of vijf generaties teruggaat, vaak slechts kleine fragmenten, soms te kort om te worden gedetecteerd.
Twee broers en zussen delen dus ongeveer 50% van hun DNA, in de vorm van vele lange segmenten, omdat ze dezelfde ouders hebben. Neven en nichten (kleinkinderen van hetzelfde echtpaar) delen gemiddeld 12,5% van hun DNA. Bij neven en nichten in de vijfde graad zijn de gemeenschappelijke segmenten veel kleiner, meer verspreid, of zelfs afwezig of onopspoorbaar.
CentimorgansDe DNA-segmenten die twee individuen delen, worden niet gemeten in fysieke eenheden, maar in centimorgans (cM). Een centimorgan komt niet overeen met een daadwerkelijke afstand op het DNA, maar met een waarschijnlijkheid van genetische recombinatie. Als vuistregel geldt dat 1 cM gemiddeld gelijk staat aan ongeveer een miljoen basen, hoewel dit getal varieert afhankelijk van de positie op het chromosoom, het betreffende chromosoom en zelfs het geslacht.
Een segment van 1 cM betekent dat er een kans van 1% is dat er op precies die locatie tijdens de meiose, het proces van de vorming van voortplantingscellen (sperma en eicellen), een recombinatiegebeurtenis (d.w.z. een uitwisseling van genetisch materiaal) plaatsvindt.
Kort gezegd komt het erop neer dat in elke generatie het DNA wordt herschikt door recombinatie tijdens de meiose. Hierdoor worden de DNA-segmenten die we van een voorouder hebben geërfd, geleidelijk aan gefragmenteerd.
Door SNP's te vergelijken, identificeren we segmenten die altijd door twee mensen worden gedeeld, wat wijst op een gemeenschappelijke oorsprong. Hun grootte, uitgedrukt in centimorgans, stelt ons in staat terug te gaan in de tijd naar deze voorouder. Hoe langer en talrijker de gedeelde segmenten (gemeten in centimorgans), hoe nauwer de verwantschap.
Op deze manier kunnen we met behulp van een paar milliliter speeksel en de analyse van honderdduizenden SNP's familiebanden reconstrueren.
DNA gebruiken om familiebanden te herstellenKortom, het is het gedetailleerde begrip van meiose en recombinatie dat onderzoekers in de genetische genealogie tegenwoordig in staat stelt om de lijn van verwantschap te traceren.
Door de grootte en de verdeling van gedeelde DNA-segmenten te analyseren, is het mogelijk om familierelaties te reconstrueren, zelfs tientallen jaren later.
Zodra het mechanisme van transmissie en fragmentatie van DNA-segmenten over generaties heen begrepen is, wordt het mogelijk om deze informatie te gebruiken om een onbekend individu te identificeren op basis van zijn of haar genetisch profiel. Dit wordt gedefinieerd door een groot aantal SNP's te analyseren.
Vergelijk het doel-SNP-profiel met die opgeslagen in genealogische databasesMaar hoe kunnen we een individu opsporen via een eenvoudig biologisch monster (bloed, speeksel, sperma, cellen) dat op een plaats delict wordt aangetroffen, als zijn genetische profiel niet in de forensische database voorkomt?
De eerste stap is het doorzoeken van andere databases naar genetische profielen die een verwantschap met het onbekende DNA aantonen. Deze databases bestaan: ze worden samengesteld uit de resultaten van tests uitgevoerd door miljoenen mensen die nieuwsgierig zijn naar hun voorouders, gezondheid of afkomst.
Om dit te doen, ontvangen deze mensen een kit, vaak online verkrijgbaar, waarmee ze hun DNA kunnen verzamelen, bijvoorbeeld door met een groot wattenstaafje over de binnenkant van hun wang te wrijven. Het monster wordt vervolgens naar een gespecialiseerd bedrijf in de Verenigde Staten gestuurd, dat de genetische gegevens analyseert. In ruil daarvoor levert het informatie op over de geografische en etnische afkomst van de persoon en kan het wijzen op mogelijke familiebanden met andere gebruikers die dezelfde test hebben gedaan.
Het zijn juist deze genetische databanken die, onder bepaalde strikte voorwaarden, door rechtshandhavingsinstanties - zoals de FBI in de Verenigde Staten - gebruikt kunnen worden om te bepalen of er in deze bestanden een SNP-profiel voorkomt dat nauw of ver verwant is aan dat van de persoon die door de politie gezocht wordt, het "doelwit".
In Frankrijk is DNA-onderzoek voor genealogische doeleinden verboden. Artikel 226-28-1 van het Franse Wetboek van Strafrecht voorziet in een boete van € 3.750 voor iedereen die DNA gebruikt voor persoonlijke of zogenaamde "recreatieve" doeleinden. Het is dus illegaal om je DNA te laten analyseren om je familiegeschiedenis te achterhalen of je stamboom te voltooien, maar in de praktijk wordt dit verbod op grote schaal omzeild. Naar schatting hebben 1,5 tot 2 miljoen Fransen al een dergelijke test ondergaan. Tot op heden is er om deze reden nog nooit een boete opgelegd. De methode is eenvoudig: bestel een testkit via een buitenlandse website, laat deze bezorgen in een ander Europees land en laat deze vervolgens terugsturen naar Frankrijk.
Genetische overeenkomstenWanneer een of meer DNA-segmenten die qua afstamming identiek zijn, worden gedetecteerd tussen het onbekende profiel van het "doelwit" en een individu dat is geregistreerd in de genetische database, betekent dit dat ze een gemeenschappelijke voorouder delen, van meer of minder veraf. Deze overeenkomsten worden in het Engels 'matches' of 'hits' genoemd – met andere woorden, compatibele genetische profielen.
Opgaande fase: teruggaan naar de vooroudersEerst traceren we de genealogie van de 'hits', de individuen waarvan het DNA voor een aanzienlijk deel overeenkomt met dat van het 'doelwit'. Het doel is om gemeenschappelijke voorouders te identificeren tussen het doelwit en deze genetische verwanten.
Wanneer hetzelfde DNA-segment door meerdere mensen wordt gedeeld, in dit geval het "doel"profiel en andere profielen, spreken we van triangulatie. Dit is een waarschijnlijk teken van een gemeenschappelijke afkomst van een gemeenschappelijke voorouder.
Voor elke genetische match is de volgende stap het bepalen van de meest recente gemeenschappelijke voorouder, de zogenaamde MRCA (Most Recent Common Ancestor). Dit is de persoon, of het echtpaar, waarvan de doelgenen en de genetische match afstammen.
Afdalende fase: het verkennen van nakomelingenZodra deze gemeenschappelijke voorouder is geïdentificeerd, veranderen we van richting en gaan we terug naar de stamboom, beginnend bij hem: we reconstrueren zijn stamboom tot op de dag van vandaag. Het doel is om al zijn nakomelingen te identificeren, dat wil zeggen de individuen die waarschijnlijk het doelwit zijn van de zoektocht.
Deze correspondentie alleen is echter niet voldoende om de gezochte persoon precies te identificeren: een MRCA kan namelijk tientallen, zelfs honderden potentiële nakomelingen hebben.
Om de zoekopdracht te verfijnen, worden verschillende genetische matches vergeleken van personen die een DNA-segment delen met de onbekende persoon, zelfs als ze niet noodzakelijkerwijs van dezelfde voorouder afstammen. Deze kruisverwijzing van informatie wordt een snijpunt genoemd. Bijzondere aandacht wordt besteed aan deze snijpunten: het zijn punten waar twee verschillende genealogische takken, verbonden met verschillende voorouders, samenkomen in hetzelfde paar of dezelfde groep nakomelingen. Deze snijpunten maken het mogelijk om het zoekgebied te verkleinen. Het is dus logischerwijs waarschijnlijk dat het "doel" in deze groep wordt gevonden.
Criteria voor het beperken van de lijst met verdachtenIn deze fase verschuift het onderzoek van puur genetische naar meer traditionele methoden. Het gaat hierbij om het onderzoeken van kandidaten om te bepalen welke het beste bij het gewenste profiel past.
Er worden verschillende criteria gebruikt om incompatibele personen te elimineren. De geschatte geografische of etnische afkomst van het DNA – bijvoorbeeld Noord-Europees, West-Afrikaans of Oost-Aziatisch – wordt gebruikt om degenen uit te sluiten die niet aan dit profiel voldoen. Ook de leeftijd van de doelpersoon wordt in aanmerking genomen. Bepaalde markers veranderen namelijk chemisch in de loop van iemands leven, wat het mogelijk maakt om de biologische leeftijd van de persoon te schatten. Jong mannelijk DNA zal dus automatisch kandidaten elimineren die te oud of te jong zijn.
Ten slotte kunnen geslacht en andere fysieke kenmerken die aan de hand van DNA worden bepaald, zoals oogkleur, haarkleur en huidskleur, ook een rol spelen bij deze selectie.
Bevestiging door een klassieke DNA-testAan de hand van verschillende screeningscriteria wordt de lijst met verdachten teruggebracht tot een klein aantal personen, of zelfs één persoon. De laatste stap is het formeel bevestigen van de identiteit door het DNA van de verdachte direct te vergelijken met het DNA dat op de plaats delict is aangetroffen.
Deze verificatie is gebaseerd op een klassieke genetische analysemethode, STR (Short Tandem Repeat) profilering, die een nauwkeurige matching van genetische profielen mogelijk maakt. Indien de persoon overleden is, is het mogelijk om zijn of haar identiteit te bevestigen door zijn of haar familieleden te testen.
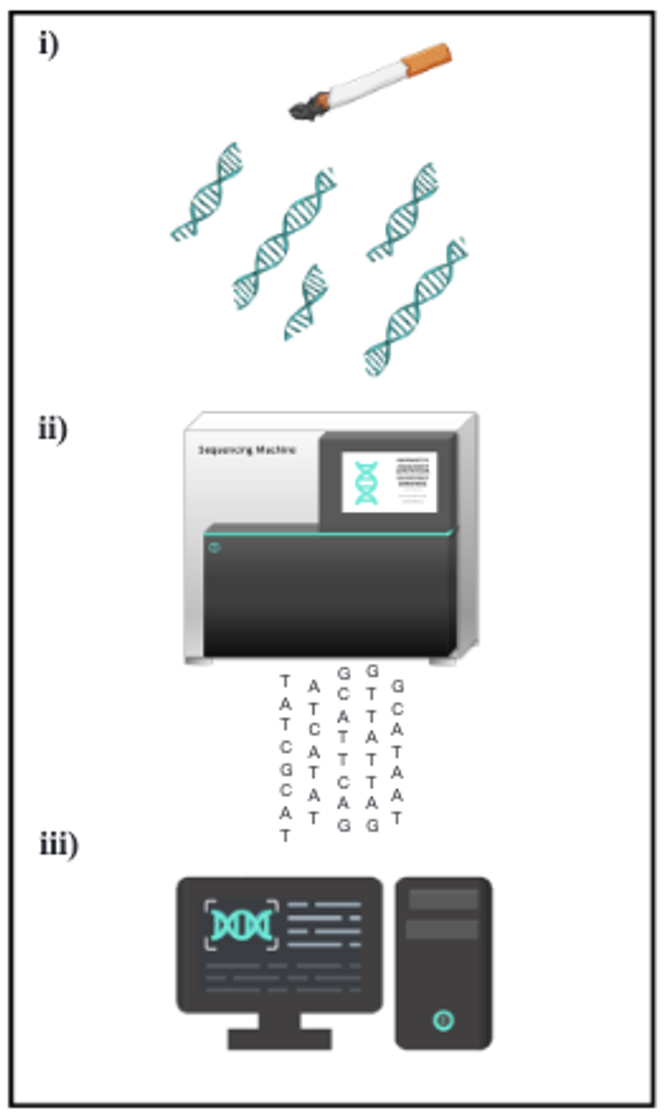
Laten we teruggaan naar de drie strafzaken waarin onderzoekers genetische genealogie gebruikten. Nadat de SNP's waren geïdentificeerd, werden de genetische profielen geüpload naar de servers van GEDmatch Pro en FamilyTreeDNA (FTDNA) om te zoeken naar mogelijke verwante profielen. GEDmatch Pro heeft ongeveer 600.000 profielen, terwijl FTDNA er meer dan 1,4 miljoen claimt – maar Noren zijn slechts licht vertegenwoordigd, wat de reikwijdte van het onderzoek in deze context beperkt.
De zoekopdrachten leverden echter te veel matches (of hits) op, waardoor de analyse lastig was. Om dit aan te pakken, ontwikkelden de onderzoekers een computerprogramma om de resultaten te filteren, matches die te frequent of niet-informatief waren weg te laten en alleen de meest relevante te behouden. Nadat het profiel door GEDmatch was gevalideerd, duurde het identificeren van matches minder dan 24 uur.
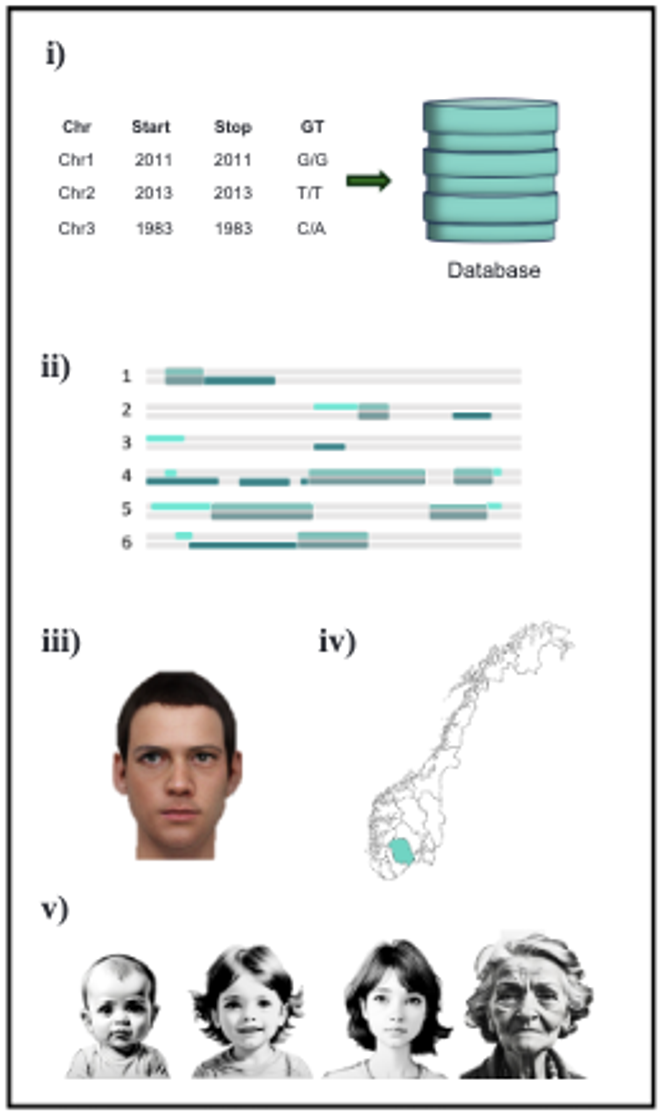 genetische evaluatie van matches (hits), voorspellingen van fenotype, afkomst
biogeografisch en leeftijd." srcset="https://img.lemde.fr/2025/07/12/0/0/261/439/320/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique -affaires-criminelles-crime-viol-meurtre-correspondances-gei-nei-tiques-hits-phei-notype-ascendance-biogei-ographique.png 320w, https://img.lemde.fr/2025/07/12/0/0/261/439/556/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-aff criminal-areas-crime-rape-murder-correspondences-gei-nei-tics-hits-phei-notype-ancestry-biogei-graphique.png 556w, https://img.lemde.fr/2025/07/12/0/0/261/439/640/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-aff criminele-gebieden-crime-rape-murder-correspondences-gei-nei-tics-hits-phei-notype-ancestry-biogei-graphique.png 640w, https://img.lemde.fr/2025/07/12/0/0/261/439/664/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-aff criminele-gebieden-crime-rape-murder-correspondences-gei-nei-tics-hits-phei-notype-ancestry-biogei-graphique.png 664w, https://img.lemde.fr/2025/07/12/0/0/261/439/960/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-aff criminal-areas-crime-rape-murder-correspondences-gei-nei-tics-hits-phei-notype-ancestry-biogei-graphique.png 960w, https://img.lemde.fr/2025/07/12/0/0/261/439/1112/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-af criminele-daden-misdaad-verkrachting-moord-correspondenties-gei-nei-tics-treft-phei-notype-ancestry-biogei-graphique.png 1112w, https://img.lemde.fr/2025/07/12/0/0/261/439/1328/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-af criminele-daden-misdaad-verkrachting-moord-correspondenties-gei-nei-tics-treft-phei-notype-ancestry-biogei-graphique.png 1328w, https://img.lemde.fr/2025/07/12/0/0/261/439/1668/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-af criminal-doings-crime-rape-murder-correspondences-gei-nei-tics-hits-phei-notype-ancestry-biogei-graphique.png 1668w, https://img.lemde.fr/2025/07/12/0/0/261/439/1992/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-af criminele-daden-misdaad-verkrachting-moord-correspondenties-gei-nei-tics-treft-phei-notype-ancestry-biogei-graphique.png 1992w, https://img.lemde.fr/2025/07/12/0/0/261/439/2301/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-af criminele-daden-misdaad-verkrachting-moord-correspondenties-gei-nei-tics-treft-phei-notype-ancestry-biogei-graphique.png 2301w" sizes="(min-width: 1024px) 556px, (min-width: 768px) 664px, 100vw" breedte="664">
genetische evaluatie van matches (hits), voorspellingen van fenotype, afkomst
biogeografisch en leeftijd." srcset="https://img.lemde.fr/2025/07/12/0/0/261/439/320/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique -affaires-criminelles-crime-viol-meurtre-correspondances-gei-nei-tiques-hits-phei-notype-ascendance-biogei-ographique.png 320w, https://img.lemde.fr/2025/07/12/0/0/261/439/556/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-aff criminal-areas-crime-rape-murder-correspondences-gei-nei-tics-hits-phei-notype-ancestry-biogei-graphique.png 556w, https://img.lemde.fr/2025/07/12/0/0/261/439/640/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-aff criminele-gebieden-crime-rape-murder-correspondences-gei-nei-tics-hits-phei-notype-ancestry-biogei-graphique.png 640w, https://img.lemde.fr/2025/07/12/0/0/261/439/664/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-aff criminele-gebieden-crime-rape-murder-correspondences-gei-nei-tics-hits-phei-notype-ancestry-biogei-graphique.png 664w, https://img.lemde.fr/2025/07/12/0/0/261/439/960/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-aff criminal-areas-crime-rape-murder-correspondences-gei-nei-tics-hits-phei-notype-ancestry-biogei-graphique.png 960w, https://img.lemde.fr/2025/07/12/0/0/261/439/1112/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-af criminele-daden-misdaad-verkrachting-moord-correspondenties-gei-nei-tics-treft-phei-notype-ancestry-biogei-graphique.png 1112w, https://img.lemde.fr/2025/07/12/0/0/261/439/1328/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-af criminele-daden-misdaad-verkrachting-moord-correspondenties-gei-nei-tics-treft-phei-notype-ancestry-biogei-graphique.png 1328w, https://img.lemde.fr/2025/07/12/0/0/261/439/1668/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-af criminal-doings-crime-rape-murder-correspondences-gei-nei-tics-hits-phei-notype-ancestry-biogei-graphique.png 1668w, https://img.lemde.fr/2025/07/12/0/0/261/439/1992/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-af criminele-daden-misdaad-verkrachting-moord-correspondenties-gei-nei-tics-treft-phei-notype-ancestry-biogei-graphique.png 1992w, https://img.lemde.fr/2025/07/12/0/0/261/439/2301/0/75/0/60063d5_upload-1-jtsljqjrtgby-gei-nei-alogie-gei-nei-tique-af criminele-daden-misdaad-verkrachting-moord-correspondenties-gei-nei-tics-treft-phei-notype-ancestry-biogei-graphique.png 2301w" sizes="(min-width: 1024px) 556px, (min-width: 768px) 664px, 100vw" breedte="664">De onderzoekers bestudeerden vervolgens de gedeelde DNA-segmenten om de segmenten te identificeren die gemeenschappelijk waren voor meerdere mensen. Deze segmenten, 'getrianguleerd' genoemd, geven aan dat dit DNA waarschijnlijk afkomstig is van een gemeenschappelijke voorouder, wat helpt bij het traceren van de familielijn.
Met behulp van de ForenSeq DNA Signature Prep Kit (Verogen), die talloze genetische markers bevat, konden onderzoekers het fenotype van de verdachte voorspellen, waaronder enkele van zijn fysieke kenmerken, zoals oog- en haarkleur, evenals zijn biogeografische afkomst, d.w.z. de regio waar zijn voorouders vandaan kwamen. Al deze markers werden geanalyseerd door middel van sequencing en vervolgens met behulp van speciale software.
Deze informatie maakte het mogelijk om bepaalde individuen uit te sluiten waarvan de kenmerken niet overeenkwamen met het voorspelde genetische profiel. In één geval bleek een zeldzame fysieke eigenschap bijzonder doorslaggevend. Zo wezen de analyses in het eerste geval op een persoon met bruine ogen, bruin haar en Europese afkomst. In het tweede geval moest de beoogde persoon bruine ogen, blond haar en ook Europese afkomst hebben gehad. Ten slotte, in het derde geval, kwam het profiel overeen met een persoon met blauwe ogen, rood haar en Europese afkomst. In beide gevallen, waarin de gewenste persoon kon worden geïdentificeerd, bleken de voorspellingen correct.
Onderzoekers maakten ook gebruik van zogenaamde genetische biogeografische analyses, waarmee de geografische afkomst van een individu kan worden bepaald aan de hand van zijn of haar DNA. Ook de commerciële sites FTDNA en GEDmatch PRO bieden dit type analyse aan.
In beide opgeloste gevallen had de gezochte persoon recente voorouders, binnen de laatste drie tot vijf generaties, die buiten Noorwegen geboren waren. Deze niet-Noorse afkomst liet voldoende duidelijke genetische sporen na om het genealogisch onderzoek effectief te kunnen sturen.
Als een van de kandidaten bijvoorbeeld een Italiaanse voorouder had, maar het DNA van de kandidaat geen bewijs van Italiaanse afkomst vertoonde, werd deze aanwijzing onwaarschijnlijk geacht. Omgekeerd, als het genetische profiel een "Centraal-Europese" component aangaf en de stamboom van een kandidaat een voorouder uit die regio onthulde, kreeg deze tak prioriteit. Dezelfde logica werd toegepast op nationaal niveau: DNA dat naar Zuid-Noorwegen wees, maakte het mogelijk om uitsluitend afstammingslijnen uit het noorden uit te sluiten. Voorzichtigheid is echter geboden, aldus de auteurs. De biogeografische analysemethoden die FTDNA en GEDmatch PRO gebruiken, zijn immers nog niet gevalideerd door peer-reviewed wetenschappelijke publicaties.
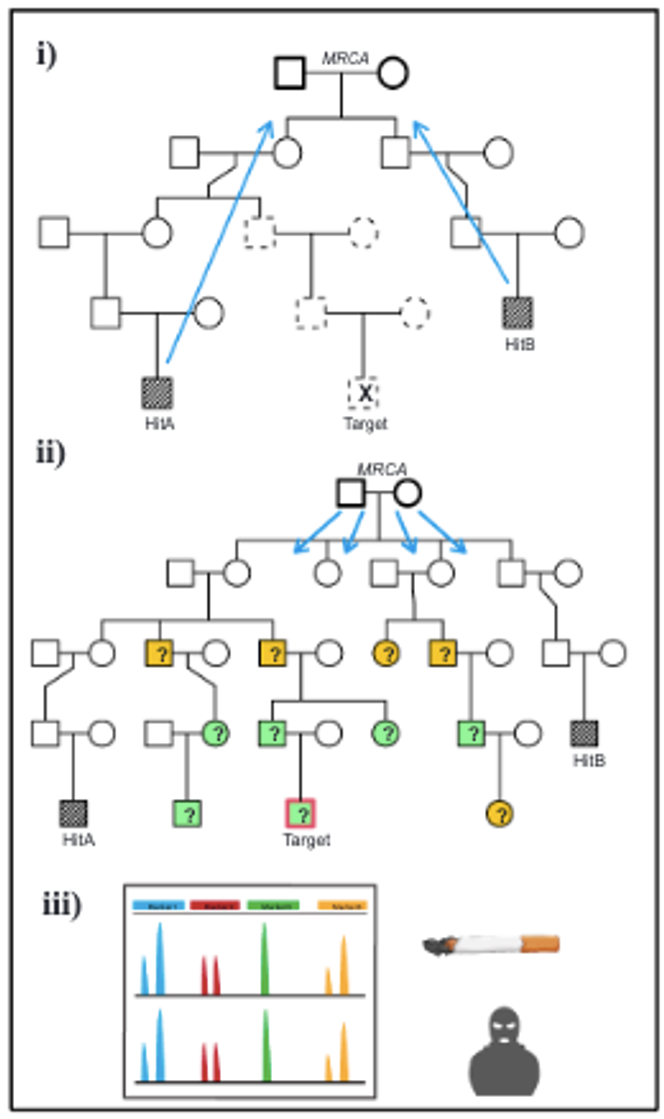 door STR-profilering van het doelwit (gezochte persoon)." srcset="https://img.lemde.fr/2025/07/12/0/0/260/438/320/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie -gei-nei-tique-affaires-criminelles-crime-viol-meurtre-ancei-tre-commun-le-plus-rei-cent-mrca-str-cible.png 320w, https://img.lemde.fr/2025/07/12/0/0/260/438/556/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-gei -nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-most-rei-hundred-mrca-str-cible.png 556w, https://img.lemde.fr/2025/07/12/0/0/260/438/640/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-gei -nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-most-rei-hundred-mrca-str-cible.png 640w, https://img.lemde.fr/2025/07/12/0/0/260/438/664/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-gei -nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-most-rei-hundred-mrca-str-cible.png 664w, https://img.lemde.fr/2025/07/12/0/0/260/438/960/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-gei -nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-most-rei-hundred-mrca-str-cible.png 960w, https://img.lemde.fr/2025/07/12/0/0/260/438/1112/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-ge i-nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-being-most-rei-cent-mrca-str-cible.png 1112w, https://img.lemde.fr/2025/07/12/0/0/260/438/1328/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-ge i-nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-being-most-rei-cent-mrca-str-cible.png 1328w, https://img.lemde.fr/2025/07/12/0/0/260/438/1668/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-ge i-nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-being-most-rei-cent-mrca-str-cible.png 1668w, https://img.lemde.fr/2025/07/12/0/0/260/438/1992/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-ge i-nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-being-most-rei-cent-mrca-str-cible.png 1992w, https://img.lemde.fr/2025/07/12/0/0/260/438/2301/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-ge i-nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-being-most-rei-cent-mrca-str-cible.png 2301w" sizes="(min-width: 1024px) 556px, (min-width: 768px) 664px, 100vw" width="664">
door STR-profilering van het doelwit (gezochte persoon)." srcset="https://img.lemde.fr/2025/07/12/0/0/260/438/320/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie -gei-nei-tique-affaires-criminelles-crime-viol-meurtre-ancei-tre-commun-le-plus-rei-cent-mrca-str-cible.png 320w, https://img.lemde.fr/2025/07/12/0/0/260/438/556/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-gei -nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-most-rei-hundred-mrca-str-cible.png 556w, https://img.lemde.fr/2025/07/12/0/0/260/438/640/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-gei -nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-most-rei-hundred-mrca-str-cible.png 640w, https://img.lemde.fr/2025/07/12/0/0/260/438/664/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-gei -nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-most-rei-hundred-mrca-str-cible.png 664w, https://img.lemde.fr/2025/07/12/0/0/260/438/960/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-gei -nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-most-rei-hundred-mrca-str-cible.png 960w, https://img.lemde.fr/2025/07/12/0/0/260/438/1112/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-ge i-nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-being-most-rei-cent-mrca-str-cible.png 1112w, https://img.lemde.fr/2025/07/12/0/0/260/438/1328/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-ge i-nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-being-most-rei-cent-mrca-str-cible.png 1328w, https://img.lemde.fr/2025/07/12/0/0/260/438/1668/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-ge i-nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-being-most-rei-cent-mrca-str-cible.png 1668w, https://img.lemde.fr/2025/07/12/0/0/260/438/1992/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-ge i-nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-being-most-rei-cent-mrca-str-cible.png 1992w, https://img.lemde.fr/2025/07/12/0/0/260/438/2301/0/75/0/9fc94bd_upload-1-j2rhbzuih9np-gei-nei-alogie-ge i-nei-tique-criminal-affairs-crime-rape-murder-ancestry-common-being-most-rei-cent-mrca-str-cible.png 2301w" sizes="(min-width: 1024px) 556px, (min-width: 768px) 664px, 100vw" width="664">Om stambomen te reconstrueren op basis van de gevonden DNA-matches, maakten de onderzoekers gebruik van verschillende bronnen: het Nationaal Bevolkingsregister, kerkregisters, volkstellingen en diverse gedigitaliseerde bronnen die beschikbaar werden gesteld door de Nationale Bibliotheek. Ze raadpleegden ook genealogische databases zoals Geni en MyHeritage, maar alleen om openbaar beschikbare genealogische informatie te benutten – zonder hun DNA-analysetools te gebruiken.
Om DNA-matches te identificeren en familiebanden te traceren, maakten onderzoekers gebruik van het Noorse Nationale Bevolkingsregister, dat gegevens bevat over geboorten, overlijdens, huwelijken, adressen en meer. Dit register is sinds 1964 gedeeltelijk en sinds de jaren negentig volledig gedigitaliseerd, met een afnemende datakwaliteit voor oudere periodes.
De parochieregisters, die tot 1942 in gebruik waren, dateren uit 1623 en dienden tevens als bevolkingsregister. Om de lacunes tussen de verschillende bronnen op te vullen, maakten onderzoekers gebruik van historische volkstellingen (met name die van 1920, 1910, 1900, 1891, 1875, 1865 en 1801), evenals niet-openbare archieven, toegankelijk via archivarissen.
De Nationale Bibliotheek heeft waardevolle ondersteuning geboden door de digitalisering van de volledige lokale krantencollecties (overlijdensberichten, biografische portretten), evenals van talrijke werken over lokale geschiedenis en genealogie. Open genealogische databases zoals Geni en MyHeritage zijn gebruikt om aanwijzingen te verkrijgen, maar hun beperkte betrouwbaarheid en betaalde toegang hebben het gebruik ervan beperkt.
Dankzij de rijkdom aan Noorse archieven (burgerlijke stand, volkstellingen, parochieregisters) konden onderzoekers de meeste inwoners van Noorwegen identificeren die een genetische link deelden met het geanalyseerde DNA en hun genealogie over vier tot zes generaties reconstrueren. De identificatie van matches die in het buitenland woonden, met name in de Verenigde Staten, bleek daarentegen complexer. De massale emigratie van Noren naar de Verenigde Staten aan het einde van de 19e en het begin van de 20e eeuw betekent dat een Noor tegenwoordig honderden verre neven en nichten aan de andere kant van de Atlantische Oceaan kan hebben.
In twee gevallen was de leeftijd van de gezochte persoon onbekend. In een ander geval viel de leeftijd binnen een nauw bereik, wat identificatie aanzienlijk vergemakkelijkte. Dit toont de interesse aan in het ontwikkelen van betrouwbare methoden om leeftijd te schatten op basis van DNA.
Opmerkelijk feit: in de twee opgeloste zaken woonden de verdachten op het moment van de feiten op minder dan 10 km van de plaats delict.
In het eerste geval waren de eerste genetische matches die via GEDmatch Pro en FTDNA werden verkregen te ver weg en onbetrouwbaar om een genealogisch onderzoek te starten: de meeste betroffen niet-Noorse personen, die zeer korte DNA-segmenten deelden, zonder duidelijke connectie. Het complexe en multi-etnische genetische profiel van de gezochte persoon compliceerde de analyse verder. Aan de andere kant kon een schatting van de geografische voorouderlijke oorsprong worden vastgesteld. Deze aanwijzingen hielpen bij het onderzoek, dat nog steeds loopt.
In het tweede geval hadden de initiële genetische matches minder dan 50 centimorgans, wat wijst op zeer verre ouderlijke banden, soms verbonden met een klein Noors dorp, met enkele connecties met de Verenigde Staten.
Met behulp van GEDmatch Pro werd een betere match gevonden, die 236 cM deelde met de gezochte persoon. Het kostte onderzoekers slechts 10 tot 15 uur om de kandidaat te identificeren na ontvangst van de resultaten van GEDmatch Pro. De geconstrueerde stamboom telde toen 319 leden, waaronder 185 levende leden.
Deze persoon had meerdere halfneven en -nichten in Noorwegen, van wie sommigen in de buurt van de plaats delict woonden. Een van hen had een consistent genetisch profiel en fenotype, inclusief gedeeltelijke Centraal-Europese voorouders.
Een STR-profieltest bevestigde dat deze persoon inderdaad de bron was van het gevonden DNA. De zaak loopt nog. Het proces heeft nog niet plaatsgevonden.
In het derde geval begon het onderzoek met FTDNA, waarbij verschillende genetische matches tussen 50 en 102 cM werden gevonden. Vier maanden later kwam er echter een grote doorbraak met een match van 87 cM op GEDmatch Pro, gekoppeld aan een Amerikaan van Noorse afkomst. Alle matches hadden voorouders uit hetzelfde Noorse dorp. De onderzoekers veronderstelden vervolgens dat er een generatiekloof van drie tot vier bestond met de gezochte persoon, waardoor het onderzoek zich op verschillende familietakken kon richten.
Er werden gerichte DNA-testen uitgevoerd. Verschillende personen gaven vrijwillig een referentie-DNA-monster af, slechts één weigerde. De verkregen resultaten maakten het mogelijk om bepaalde familietakken uit te sluiten en het onderzoek te richten op een tak van halfneven en -nichten.
Uit het onderzoek blijkt dat de gezochte persoon is overleden. De schuldige partij kan dus nooit worden berecht voor het gepleegde feit. Het blijkt dat hij een bekende was bij de politie en dat zijn DNA in hun administratie stond. Dit werd onderworpen aan STR-profilering, een methode die bestaat uit het analyseren van specifieke regio's van het genoom, die van individu tot individu variëren, om mogelijke relaties vast te stellen. Het verkregen genetische profiel kwam overeen met dat van het DNA dat op de plaats delict werd aangetroffen.
Tegen de tijd dat de zaak werd opgelost, hadden de onderzoekers in een jaar tijd tussen de 2.000 en 3.000 uur aan werk besteed en de stamboom bevatte 3.785 personen, waaronder 1.656 nog in leven.
De Noorse ervaring is geen uitzondering. Zeer recentelijk heeft genetische genealogie het in Canada mogelijk gemaakt een zeventien jaar oude cold case op te lossen. Op 17 september 2025 maakte de Montreal City Police Service (SPVM) bekend dat zij dankzij deze techniek de moord op een 26-jarige jonge vrouw, die in 2008 in haar appartement werd vermoord, had opgelost. DNA-analyses leidden tot de identificatie van de dader: een man die in 2021 overleed in een detentiecentrum, waar hij een straf uitzat wegens diefstal en poging tot moord. Er waren geen familiebanden die hem met het slachtoffer verbonden. Hij zou een paar dagen voor de tragedie contact met haar hebben opgenomen, nadat ze haar auto te koop had aangeboden op een online advertentiesite.
Ethische, juridische, maatschappelijke kwestiesDe afgelopen jaren hebben talloze onderzoeken zich gericht op het gebruik van genetische gegevens in de context van genetische genealogie voor onderzoeksdoeleinden. Ze onderzochten verschillende gevoelige kwesties: technische vooruitgang, maatschappelijke kwesties (met name respect voor privacy), publieke zorgen – of het nu gaat om toestemming, informatie of vrijwillige deelname aan gerichte DNA-tests –, zonder de juridische uitdagingen te vergeten die deze methode met zich meebrengt voor de rechtshandhaving.
Om publieke steun te krijgen en misbruik van genetische genealogie te voorkomen, is het essentieel om strikte richtlijnen te volgen die bepalen wanneer en hoe deze methode moet worden gebruikt, zeggen Noorse onderzoekers. "Zoals bij elk werk waarbij gebruik wordt gemaakt van gevoelige gegevens, is handelen met professionaliteit en integriteit jegens de betrokkenen essentieel. Strikte naleving van deze principes, gecombineerd met voortdurende wetenschappelijke vooruitgang, zal doorslaggevend zijn voor de implementatie en acceptatie van deze aanpak in de meest serieuze strafrechtelijke onderzoeken", concluderen zij.
Voor meer informatie:
Aanes H, Vigeland MD, Star B, et al. Heating up three cold cases in Norway using investigative genetic genealogy . Forensic Sci Int Genet. 2025 Mar ;76 :103217. doi : 10.1016/j.fsigen.2024.103217
Wang M, Chen H, Luo L, et al. Forensic investigative genetic genealogy : expanding pedigree tracing and genetic inquiry in the genomic era . J Genet Genomics. 2025 Apr ;52(4) :460-472. doi : 10.1016/j.jgg.2024.06.016
Guerrini CJ, Robinson JO, Elsaid MI, et al. FIGG at 5 : An update on US public perspectives on forensic investigative genetic genealogy five years after its introduction to criminal investigations . Forensic Sci Int. 2025 Feb ;367 :112372. doi : 10.1016/j.forsciint.2025.112372
Tuazon OM, Wickenheiser RA, Ansell R, et al. Law enforcement use of genetic genealogy databases in criminal investigations : Nomenclature, definition and scope . Forensic Sci Int Synerg. 2024 Feb 8 ;8 :100460. doi : 10.1016/j.fsisyn.2024.100460
Gurney D, Press M, Moore C, et al. The need for standards and certification for investigative genetic genealogy, and a notice of action . Forensic Sci Int. 2022 Dec ;341 :111495. doi : 10.1016/j.forsciint.2022.111495
Glynn CL. Bridging Disciplines to Form a New One : The Emergence of Forensic Genetic Genealogy . Genes (Basel). 2022 Aug 1 ;13(8) :1381. doi : 10.3390/genes13081381
Dowdeswell TL. Forensic genetic genealogy : A profile of cases solved . Forensic Sci Int Genet. 2022 May ;58 :102679. doi : 10.1016/j.fsigen.2022.102679
Tillmar A, Fagerholm SA, Staaf J, et al. Getting the conclusive lead with investigative genetic genealogy - A successful case study of a 16 year old double murder in Sweden . Forensic Sci Int Genet. 2021 Jul ;53 :102525. doi : 10.1016/j.fsigen.2021.102525
Wickenheiser RA. Forensic genealogy, bioethics and the Golden State Killer case . Forensic Sci Int Synerg. 2019 Jul 12 ;1 :114-125. doi : 10.1016/j.fsisyn.2019.07.003
Bijdragen
Hergebruik deze inhoudLe Monde





